图的基本定义
设$G=(V,E)$是一个无向图,其中顶点集$V={v_1,…,v_n}$,对于一个无向图而言,$w_{ij}=w_{ji}$。一个顶点的$v_i\in{V}$的按照如下定义:
定义一个指示向量,$\mathbb{1}_A={f_1,…,f_n}’$,当向量$v_i\in A$时,$f_i=1$,否则$f_i=0$,对于两个不相交的集合,定义:
对于一个顶点集合$A\subset V$的大小有如下两种定义:
$A$中所包含顶点的数量
图的拉普拉斯矩阵以及它的的特性
未归一化的图拉普拉斯矩阵
未归一化的拉普拉斯矩阵定义如下:
特性1. 对于每个向量$f\in R_n$,可以得到:
证明:通过$d_i$的定义,可以得到:
特性2. $L$是对称,半正定矩阵:
$L$的对称性来源于$W,D$,半正定矩阵来自于特征1,对于任何向量都满足$f’Lf\ge 0$.
特性3. L最小的特征值是0,对应的特征向量是$\mathbb{1}$
由$f’Lf=f’\lambda f\ge 0$,且$f’f\ge 0$,可得$\lambda\ge 0$
特性4. L有n个非负实特征值,$\lambda_n\ge…\lambda_2\ge\lambda_1\ge 0$
很明显
特征5. 假设G是一个非负权重无向图,那么$L$特征值0的多重性$k$等同于在该图中所包含的连通图的个数,对应的0特征值的特征向量等于这些连通图的指示向量
证明: 首先假设$k=1$,$f$是该特征值的特征向量,那么可以得知,由于$w_{ij}$都是非负值,所以当两个顶点相连时,则${(f_i-f_j)}^2$必须为0,即$f_i = f_j$。因此,在图中所有顶点只要互相连接,那么$f$对于这些顶点而言,就必须是常量。因此若一个无向图中存在一个连通图,那么$f$在该连通图上为常量,最终可以得到一个常数向量$\mathbb{1}$作为特征值0的特征向量,它同时也是该联通图的指示向量。
假如顶点按照他们所属的联通图排列,不难想象他们的邻接矩阵$W$是一个块对角的形式,同样的$L$矩阵也是。
其中的每个块$L_i$都是一个拉普拉斯矩阵,对应的是第$i$个连通图。由于它是块对角矩阵,所以$L$的频谱是$L_i$谱的并集,$L$的特征向量就是$L_i$的特征向量,其它块的位置对应为$0$。因此,$L$有许多特征为0的特征值,并且0特征值对应的特征向量为相应的连通图的指示向量。
归一化的图拉普拉斯矩阵
通常文献中有两种形式的图归一化矩阵,两种矩阵互相关联,定义如下所示:
其中第一个$L_{sym}$是对称矩阵,而第二个$L_{rw}$是随机游走矩阵,相对于未归一化的拉普拉斯矩阵而言,归一化的拉普拉斯矩阵具有如下类似的性质。
特性1. 对于每个向量$f\in R_n$,可以得到:
证明类似于未归一化的拉普拉斯矩阵,此处不再赘述。
谱聚类算法
unnormalized spectral clustering
normalized spectral clustering

实验
unnorm knn graph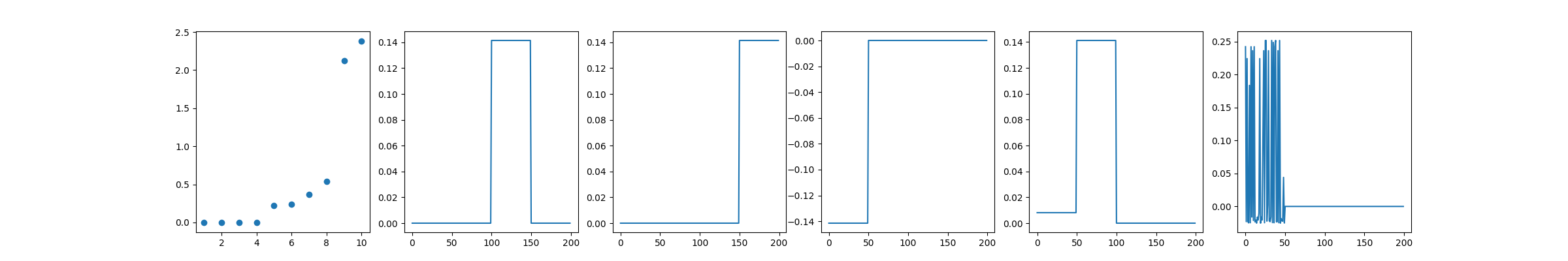
unnorm knn graph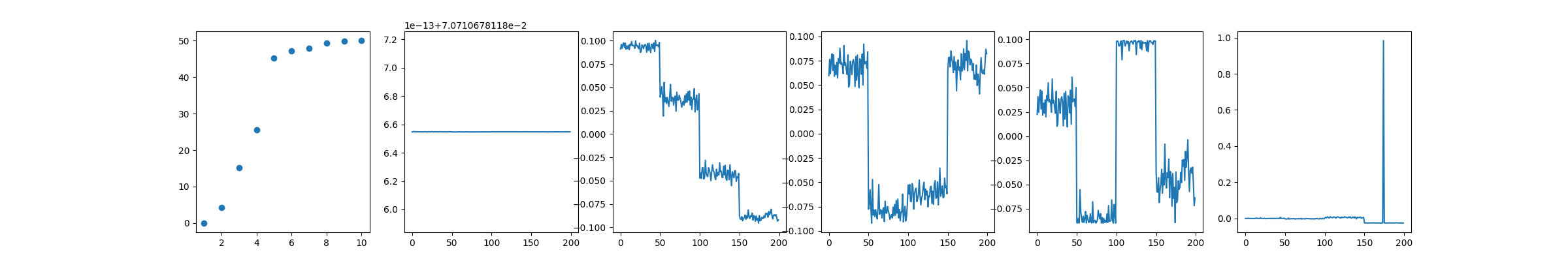
程序
# -*- coding: utf-8 -*-
# @Time : 2019/10/2 19:38
# @Author : zhao pengfei
# @Email : zsonghuan@gmail.com
# @File : spectral_clustering.py
import numpy as np
import matplotlib.pyplot as plt
import seaborn as sns
import matplotlib
matplotlib.use("TkAgg")
def plot_value_vector(value, vector):
plt.figure(figsize=(24, 4))
plt.subplot(1, 6, 1)
plt.scatter(range(1, 11), value[:10])
plt.subplot(1, 6, 2)
plt.plot(vector[:, 0])
plt.subplot(1, 6, 3)
plt.plot(vector[:, 1])
plt.subplot(1, 6, 4)
plt.plot(vector[:, 2])
plt.subplot(1, 6, 5)
plt.plot(vector[:, 3])
plt.subplot(1, 6, 6)
plt.plot(vector[:, 4])
plt.show()
#generate gaussian dataset
y1 = np.random.normal(2, 0.2, 50)
y2 = np.random.normal(4, 0.2, 50)
y3 = np.random.normal(6, 0.2, 50)
y4 = np.random.normal(8, 0.2, 50)
y = np.hstack((y1, y2, y3, y4))[:,None]
# plt.hist(y, bins=300)
# plt.title("Histogram of the sample")
# plt.show()
data = np.repeat(y, 200, axis=1)
data_t = data.T
#full graph unnorm
W = np.exp(-1 * (np.abs(data - data_t) ** 2) / 2)
#self circle removed
W = W - np.eye(W.shape[0])
D = np.zeros_like(W)
for i in range(W.shape[0]):
D[i, i] = np.sum(W[i, :])
L = D - W
eigenvalue,featurevector=np.linalg.eig(L)
sort_eigenvalue_index = np.argsort(eigenvalue)
sorted_value = eigenvalue[sort_eigenvalue_index]
sorted_vector = featurevector[:, sort_eigenvalue_index]
plot_value_vector(sorted_value, sorted_vector)
#knn graph unnorm
W = np.exp(-1 * (np.abs(data - data_t) ** 2) / 2)
#self circle removed
W = W - np.eye(W.shape[0])
for i in range(W.shape[0]):
row = W[i, :]
sort_index = np.argsort(row)
knn_value = W[i, sort_index[-10:]]
knn_index = sort_index[-10:]
new_value = np.zeros_like(row)
new_value[knn_index] = knn_value
W[i, :] = new_value
D = np.zeros_like(W)
for i in range(W.shape[0]):
D[i, i] = np.sum(W[i, :])
L = D - W
eigenvalue,featurevector=np.linalg.eig(L)
sort_eigenvalue_index = np.argsort(eigenvalue)
sorted_value = eigenvalue[sort_eigenvalue_index]
sorted_vector = featurevector[:, sort_eigenvalue_index]
plot_value_vector(sorted_value, sorted_vector)